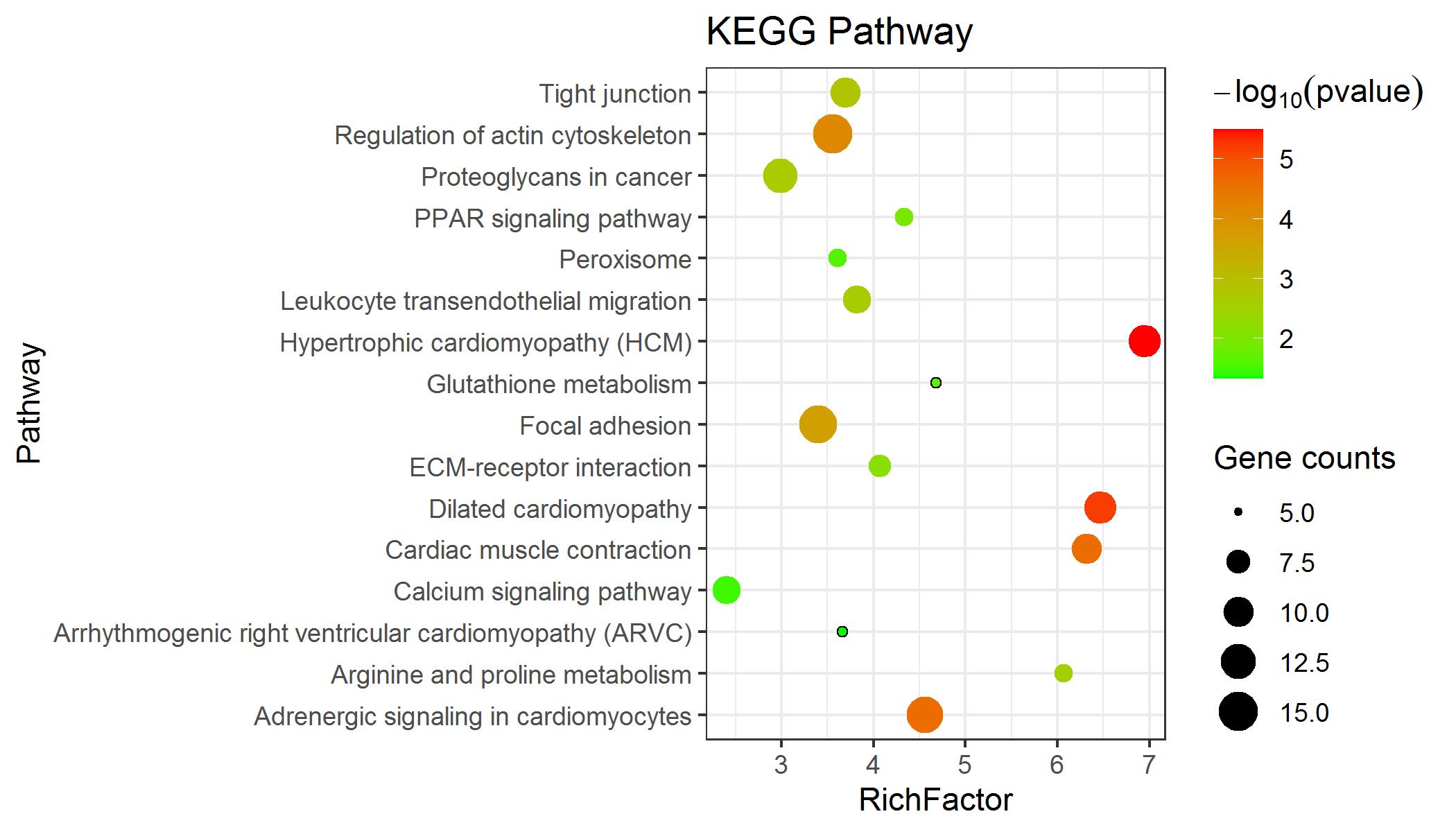
GO与KEGG富集分析可视化之气泡图绘制
写在前面
这是一篇利用ggplot2将DAVID功能富集分析的结果可视化为气泡图的学习笔记。
1 数据说明
从DAVID中导出的功能富集分析数据,主要包括Term、Count、%、pvalue、Genes、Fold Enrichment、Benjamini和FDR。气泡图由x轴、y轴、气泡的大小和气泡的颜色组成,其中x轴通常为Gene Ratio或Rich Factor,y轴为Term,气泡的大小用Count表示,气泡的颜色用p-value、q-value、adjusted p-value等表示。
-
DAVID结果中的
%表示involved genes/total genes,即Gene Ratio。 -
DAVID结果中的
Fold Enrichment表示Rich Factor。Fold Enrichment = (k/n)/(M/N),Rich Factor = k/M,而n与N为恒量,即Fold Enrichment = Rich Factor*C,C=N/n为常数。故Flold Enrichment与Rich Factor是同一个东西。
-
在统计学中,FDR(False Discovery Rate)表示在所有R次拒绝零假设中错误发现的期望比例。FDR校正后的p-value值,即q-value。因此一般可认为FDR = q-value = adjusted p-value。
在所有从p-value估算q-value的方法中,Benjamini & Hochberg法(BH法)应用最广泛。BH法关于q-value的计算公式为:
q-value = p-value*(m/k)
其中,m为检验的次数,k为本次检验的p-value在所有检验中的秩。
2 代码
library(ggplot2)
pathway = read.table("C://Users/Administrator/Desktop/Data.csv",header=T,sep=",")
p = ggplot(pathway,aes(RichFactor,Pathway)) #Pathwy即Term。
p = p + geom_point()
p= p + geom_point(aes(size=GeneCounts)) # 用GeneCounts表示气泡的大小
pbubble = p + geom_point(aes(size=GeneCounts,color=-1*log10(pvalue)))
pbubble = pbubble + scale_colour_gradient(low="green",high="red")# 自定义渐变颜色
# 绘制pathway富集散点图
pr = pbubble + scale_colour_gradient(low="green",high="red") + labs(color=expression(-log[10](pvalue)),size="Gene counts",x="RichFactor",title="KEGG Pathway")
pr = pr + theme_bw() # 改变图片的样式(主题)
ggsave("C://Users/Administrator/Desktop/KEGG Pathway.pdf",height=4) # 保存为.pdf格式
ggsave("C://Users/Administrator/Desktop/KEGG Pathway.jpg",height=4) # 保存为.jpg格式
ggsave("C://Users/Administrator/Desktop/KEGG Pathway.eps",height=4) # 保存为.eps格式
#可用ggsave("C://Users/Administrator/Desktop/KEGG Pathway.jpg",width=4,height=4)设定画布的大小
【Tips】
- 关于
read.table()函数,数据文件为.csv格式,故其参数为sep=","而非sep="\t"。sep="\t"用于.tsv格式的数据。 - 如果Term较短,则气泡图整体较宽,可在原始数据中的Term前加空格。
(按道理可以用代码实现这一操作,待后续学习)
3 结果